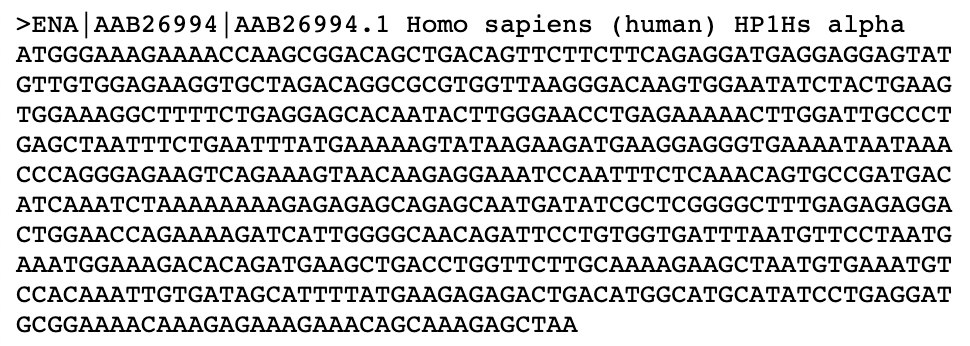
01 设计质粒
2). Amp- T7pro-GST-Thrombin-Insert-TAA- T7term -Lac
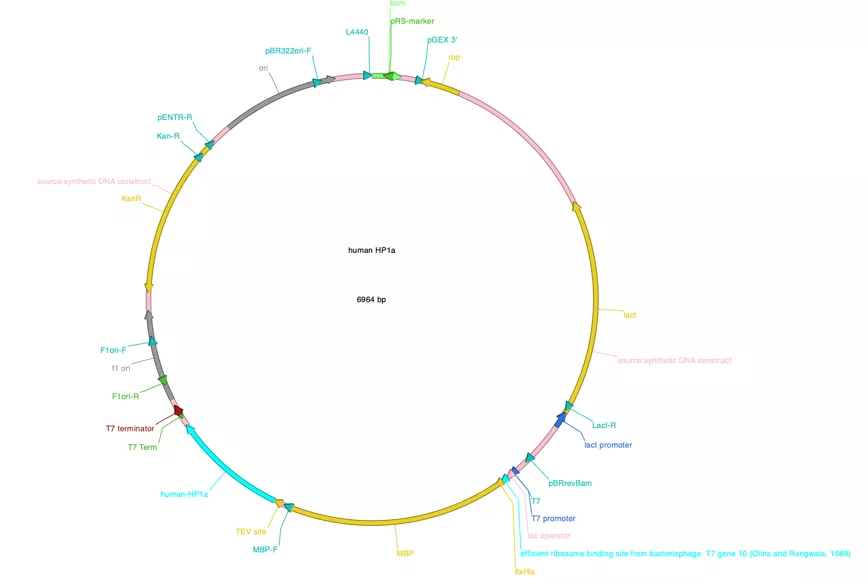
图一. Addgene pMJ806 (由Jennifer Doudna提供)的环形图,以及人源全长HP1a.
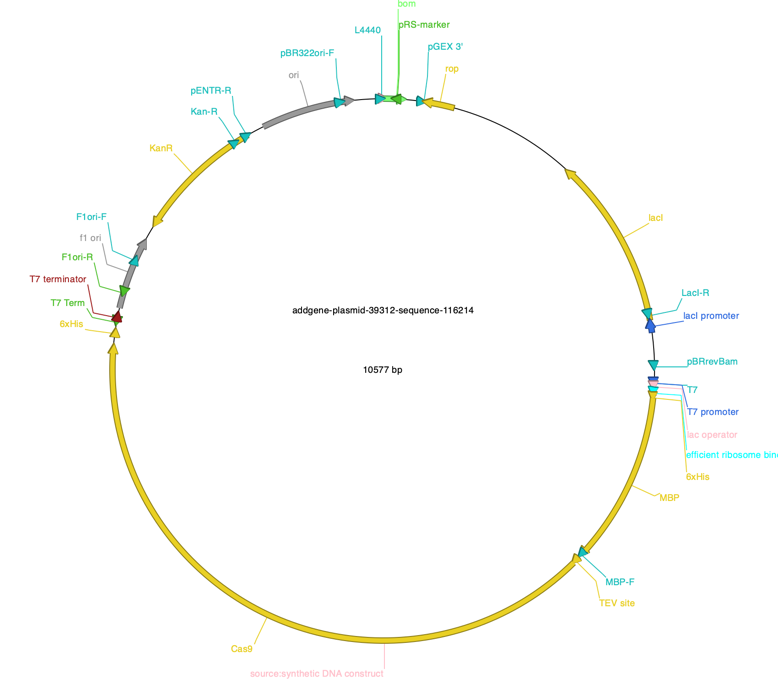
进行拼接后,我们获得了表达人源全长HP1a的目的质粒(纸面上的)。其环形图如下:
图二. 构建的目的质粒环形图,包含有Kan-T7pro-his6-MBP-Tev-humanHP1a-T7term-Lac,全长为6964bp (insert, 576bp; template 6388 bp)。
02 设计引物
图三. LIC的引物设计。
从上图可以看出,LIC的引物设计是非常严苛的,呆板的。载体的引物位点是严格控制的,一半以终止密码为起始,包含~20个碱基对;另一半在蛋白酶切割位点处。Insert的上下游引物都必须包含一部分template,一部分insert的碱基序列,因此相对较长,~35bp。在50 bp内,IDT的DNA合成准确率是可以保证的,因此不必担心含有突变。
03 订购引物,准备PCR的模板
04 为第二天的PCR和LIC反应做准备
小结